
LIGAMIENTO
Describe el fenómeno por el que alelos en genes vecinos, ubicados en el mismo cromosoma, serán transmitidos juntos más frecuentemente que por azar (Curtis, 2006). La posibilidad de segregar independientemente es directamente proporcional a la distancia entre los genes, es decir, entre más separados dentro de un cromosoma se encuentren dos genes, más probabilidad tendrán de segregar independientemente. Drosophila tiene cuatro grupos de ligamiento correspondiente a sus cuatro cromosomas, tal como se observa en la imagen 12 (Matta 2010). Los genes w y B según el mapa de ligamiento segregan de manera independiente, ya que la distancia entre ellos es de 56.1 cM. No obstante, los genes w y y se encuentran a una distancia de 1.5 cM, por lo tanto se encuentran ligados entre ellos.

Imagen 12: Mapa de ligamiento de D. melanogaster señalando los genes usados (Bar, Yellow, White). (Tomado de http://slideplayer.es/slide/4722157/).
Ejemplo, se cruzan tres mutantes, uno con cuerpo negro ( b ), otro con ojos purpura ( p ) y por ultimo uno con alas curvadas ( c ), siendo los tres recesivos respecto a la cepa silvestre.
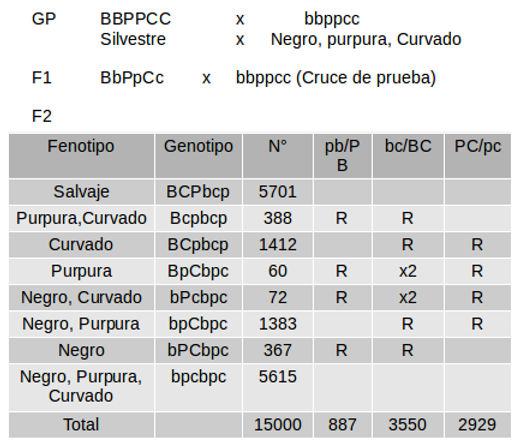
Imagen 13. Cruce para ligamiento de tres puntos. Las frecuencias de los parentales son aquellas que presentan mayor valor, respecto a las demás.
Frecuencia de recombinación: Es proporcional a la distancia física entre genes. Se usa para crear mapas de ligamiento. Se obtiene dividiendo el número de recombinantes en el numero total. Su unidad son los centimorgan (cM) (Curtis, 2006).


Imagen 14. Mapa de ligamiento para tres puntos.
Coeficiente de coincidencia (COC): Mide si los entrecruzamientos son independientes entre si (Curtis, 2006).
Coeficiente de interferencia (COI): Es la reducción de la cantidad de los dobles recombinantes esperados y el indice entre los dos dobles recombinantes observados (Curtis, 2006).


